El establecimiento de hipótesis
en los casos de parentesco también tiene sus matices. Ya vimos en los
comentarios sobre el significado del LR
(http://parentela.familias.name/#category8) que es posible que no conozcamos
todos los escenarios posibles de nuestros casos. Como ya vimos, el LR no nos dice si una hipótesis es cierta o
no, más bien, si los resultados apoyan más una hipótesis que otra. Pero resulta
que ambas hipótesis podrían no ser ciertas!!
Imaginaros que una mujer que
viaja con tres menores (niña1, niña2 y niño3) declara en la frontera que son
sus hijos. La policía detecta que el pasaporte de la mujer es falso. Esto
podría ser un caso de inmigración ilegal, pero también pude ser algo más grave
como tráfico de menores. Para aclarar la situación os piden que hagáis estudios
de ADN con el fin de determinar si los tres menores son hijos de esta mujer…
y tras analizar aSTRs veis que la niña1 presenta incompatibilidades con esta
mujer en 12 de los 21 marcadores analizados y los otros dos menores son compatibles
con la mujer.
Bueno, pues parece un caso fácil.
Y si calculamos LRs suponiendo «maternidad» vs. «no
relacionado» para cada uno de los tres menores obtendríamos un LR muy
cercano a 0 en el caso de la niña1 (demasiadas mutaciones para que la hipótesis
de maternidad sea cierta!) y LRs muy elevados en el caso de los otros dos
menores.
Hasta aquí todo perfecto, pero en los casos en los que se vean
involucrados varios individuos y tengamos exclusión en uno de ellos, os
recomiendo ir un poco más allá y pensar en la posibilidad de otras hipótesis.
Tenemos claro que la niña1 no es hija de esta mujer, pero quizás esté
emparentada por vía paterna con los otros dos menores o al menos con uno de
ellos. También tendremos que ver entonces si la niña2 y el niño3 pueden tener o
no tener el mismo padre, con las siguientes hipótesis:
H1 = la niña2 y el niño3 son hermanos completos, la mujer es su madre
H2 = la niña2 y el niño3 son medio-hermanos, la mujer es su madre
Imaginaros que obtenemos esto cuando hacemos este cálculo con
Familias:

Claramente, H1 se ve favorecida
por los resultados (LRH1vsH2 = 42e+06). Así que ahora podemos
plantear las siguientes hipótesis, pero sin perder de vista que lo que estamos
intentando aclarar es si la niña1 puede pertenecer a este grupo familiar de
alguna forma o se trata de una persona no emparentada con ninguno de ellos.
Planteemos entonces las hipótesis:
H3 = la niña2 y el niño3 son hermanos completos (la mujer es su madre),
y la niña 1 es medio hermana paterna de 2 y 3.
H4 = la niña2 y el niño3 son hermanos completos (la mujer es su
madre), y la niña 1 no está relacionada con este pedigrí.
Quizás el LR aquí no sea muy elevado (imaginaros que alrededor de 380)
y decidamos ampliar análisis. El estudio de XSTRs nos ayudaría mucho en este
caso, está clarísimo. Imaginaros que hemos analizado las 4 muestras, que la
niña1 y la niña2 comparten alelos en cada cluster y que planteamos las mismas
hipótesis H3 y H4. Con FamLinkX podemos calcular de manera muy sencilla si los
resultados de nuestro análisis de XSTRs apoyan más una hipótesis que otra.
Supongamos que obtenemos LRH3vsH4 = e+07. Menos mal que hemos
pensado en otras hipótesis y no nos hemos limitado a informar de la exclusión
entre la niña1 y la madre!! Está claro
que las consecuencias podrían haber sido terribles para esta madre.
Pero Thore me ha dicho que hay una forma mejor de hacer los cálculos!
No es necesario que bombardeemos con tantos LRs en los informes… se pueden contrastar
todas las hipótesis a la vez:
H1 = la niña2 y el niño3 son hermanos completos, la mujer es su madre
H2 = la niña2 y el niño3 son medio-hermanos, la mujer es su madre
H3 = la niña2 y el niño3 son hermanos completos (la mujer es su
madre), y la niña 1 es medio hermana paterna de 2 y 3.
H4 = la niña2 y el niño3 son hermanos completos (la mujer es su
madre), y la niña 1 no está relacionada con este pedigrí.
Si hacemos esto con Familias (marcadores autosómicos) y escalamos en
H4, obtenemos:

Es decir, un LR de unos 380 favoreciendo H3: lo mismo que cuando
comparábamos H3 y H4 sólo. Veis que para H1 nos sale 1 (como H4 escalado). Es
porque H1 y H4 son básicamente la misma hipótesis, pero la hemos definido para
poder compararla con H2. Para ello, escalamos en H2 y obtenemos:
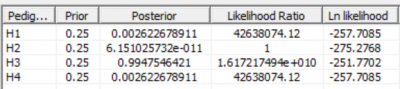
En este caso, H1 se ve favorecida 42E+06 veces, como ya habíamos visto
al comparar sólo H1 y H2. De nuevo, H1 y H4 nos da el mismo resultado porque
son en realidad la misma hipótesis.
Bueno, pues espero que os haya sido útil este ejemplo! Nos ha servido
para ilustrar una vez más lo importante que son las hipótesis y tener la mente
abierta a varias posibilidades!!


Mi querida Claudia!
Muchas gracias por tu comentario! La verdad es que yo no sé que haría sin los XSTRs en algunos casos! Son fundamentales en ciertos tipos de parentesco. Es verdad que no hya muchos estudios poblacionales, pero es tan fácil hacerlos!!
Abrazos!
Querida Lourdes, sin duda un EXCELENTE post!!! Muchas gracias por acordarte de mi querido cromosoma X. Creo que todavía es muy complicado que los investigadores se acuerden que existe otra herramienta a parte de los autosómicos, y que hay casos donde H1 y H2 con los aSTR no se pueden distinguir. Creo que es porque ya tienen implantado en sus labs todo el mecanismo de estudio con los aSTRs y les parece más fácil aumentar el numero de marcadores autosómicos, que utilizar el cromosoma X. Claro está que hay un fallo enorme en el estudio de X-STRs, que es la falta de bases de datos poblacionales. Son pocos los países que ahora mismo tienen bases de datos de X-STRs.
Un abrazo a todos
Gracias mi querida Mari! Tu sabes de sobra los problemas que generan los parentescos:)))) Me alegra mucho que sea útil!! Siempre tan generosa conmigo. Besitos desde un Madrid muy invernal!
Oh, me encantó esta discusión… Hay muchos casos en que tenemos estos problemas. Encuentro que es un excelente ejercicio para aplicar en todos nuestros casos. Muchas Gracias Lourdes! Saludos desde Chile