Ya os he hablado de Magnus Vigeland (ver https://parentela.familias.name/category/category4/). Pues bien, él ha desarrollado mucho software en R, y ahora Familias puede aprovecharse de ese trabajo!
Thore me ha enviado un ejemplo en el que se demuestra claramente el potencial que tiene combinar Familias y R. Podemos leer en R un archivo de Familias (.fam) con el comando «readFam», y también podemos dibujar los pedigríes definidos en el archivo de Familias con el comando «plotPedList».
Para poder hacerlo primero tenéis que saber dónde debe estar almacenado el archivo .fam para que R lo pueda leer. Para esto sólo tenéis que teclear en R:
getwd()
Y R os responderá dónde tenéis que almacenar el archivo .fam (también podríais indicar una ruta, pero es más fácil almacenar el archivo en el working directory de R).
Bueno, pues vamos a ver el ejemplo de Thore usando el archivo de familias sibs3.fam que podéis descargar aquí (ya sabéis, teclas Ctrl + S para descargarlo) y que debéis guardar en el working directory.
Ahora tenéis que instalar el paquete de R para aplicaciones forenses llamado «forrel». Para instalarlo sólo debéis teclear lo siguiente en Rstudio o en la consola R (se necesita conexión a internet):
install.packages(«forrel»)
La instalación de forrel sólo hay que hacerla una vez.
Ok, ahora ya estamos preparados para el ejemplo. Debéis teclear en R:
library(forrel)
x = readFam(«sibs3.fam», verbose = F)
plotPedList(x, marker = 1, shaded =
typedMembers, frametitles = names(x),
newdev = T, dev.width = 8)
Y en seguida veréis este resultado:
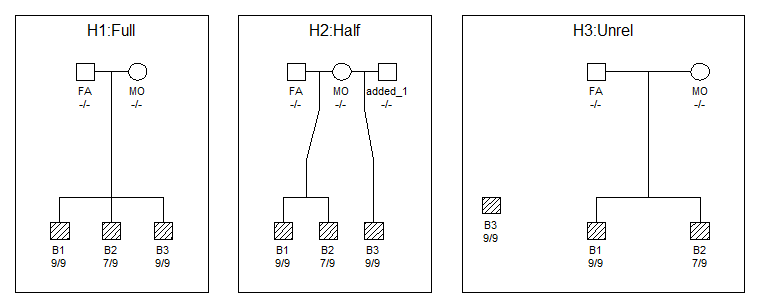
Maravilloso!! Muy útil para incluir los pedigríes en nuestros informes!!
También podemos calcular directamente los LRs en R si además tecleamos:
kinshipLR(x, ref = 3)
Y obtendremos:
## Total LR:
## H1:Full H2:Half H3:Unrel
## 8.333333 2.916667 1.000000
En el comando anterior, ‘ref = 3’ significa que los LRs están escalados con respecto a H3, es decir os da el LR con ‘H3:Unrel’ como referencia. Podéis encontrar más documentación y ejemplos si tecleáis:
?kinshipLR
Si decidís usar el working directory, cada vez que queráis usar esta herramienta con otro archivo, solo debéis modificar el nombre del archivo en las líneas de comandos de arriba:
En lugar de x = readFam(«sibs3.fam», verbose = F), debeis escribir x = readFam(«nombre de vuestro archivo.fam«, verbose = F)
O si preferís, en lugar de guardar el archivo .fam en el working directory de R, también podríais indicar la ruta en donde lo tengáis guardado, por ejemplo:
x = readFam(«C:/Users/lourditasmt/Documents/sibs3.fam», verbose = F)
Que rápido se hace y que bonito queda!!
Magnus y Thore, mil gracias, sois genios!!


Genial speedy Thore! Mil gracias!!
Y muchas gracias también a Magnus por actualizar pedsuit software en un futuro!
Tenemos mucha suerte con vosotros!!
Aprovecho para deciros que el símbolo (“) de las lineas de comando puede dar problemas si se escribe como («) . Me han comentado que algunos usuarios del blog ven « en lugar de “. Hay que cambiar « por “ si lo veis así.
Hola Cecilia!
I have tried to explain how the approach Lourdes explained can be modified to deal with X-chromosomal markers:
http://familias.name/X-Files.pdf
Hola Cecilia!
Muchísimas gracias por tu pregunta!
Pues no es necesario dibujar los pedigríes para calcular el LR, pero sí que es muy útil porque te permite comprobar que tus hipótesis son correctas.
En principio esto es para archivos .fam (para Familias, no para FamLinkX), pero por si acaso he probado con un archivo .sav, y no me funciona. Quizás cambiando algo en el código sí que funcione, así que le he preguntado a Thore y nos contestará en cuanto pueda.
Desde luego sería genial si funcionara con archivos .sav, pues a mi me pasa lo mismo que a ti; cuando tengo que crear pedigríes no descritos en FamLinkX, siempre tengo que repasarlos mucho para ver que no me he equivocado.
Bueno, pues quizás el lunes tengamos respuesta!!
Genial tu pregunta! Thanks!!
Estimada Lourdes, una pregunta: hacer el grafico en R es necesario para hacer los CALCULOS en Fam Link X ? La pregunta es porque estamos trabajando en un caso bastante complejo y el gráfico del pedigree no esta dentro de los propuestos en «Fam link X» por lo que estamos cargando las relaciones mediante «create pedigree». Es un caso bastante complicado y no estamos seguros de que el resultado que arroja el programa es correcto.
Si esta dentro de las posibilidades lo podriamos enviar a fin de recibir alguna ayuda – Desde ya muchas gracias!!!
fantastico¡¡ miles de gracias a los tres¡¡ muy muy util